NEWS
新闻资讯
|
项目文章 | H3K79甲基化通过增加光合作用促进高光强下太平洋亚历山大藻的快速生长
发表单位:中国海洋大学海洋遗传育种教育部重点实验室 发表日期:2023年6月11日 期 刊:Science of the Total Environment(IF:10.753) 2023年6月11日中国海洋大学海洋遗传育种教育部重点实验室在Science of the Total Environment(IF:10.753)发表了题为“H3K79 methylation promotes rapid growth of Alexandrium pacificum under high light intensity via increased photosynthesis”的研究论文。该研究结果表明H3K79me在调节太平洋亚历山大藻的快速生长中发挥了作用,光合作用可能是一个重要的调节途径,这是首次从H3K79me的角度为有毒赤潮的形成提供了表观遗传学证据。爱基百客为该研究提供ChIP-seq的技术支持。
太平洋亚历山大藻是造成有害藻华的主要物种之一,在一定范围内,增加光照强度可以促进太平洋亚历山大藻的快速生长。因此,有必要阐明太平洋亚历山大藻爆发式增长的内在机制。先前的研究发现了调控太平洋亚历山大藻生长的多种修饰类型,尤其是组蛋白H3赖氨酸79的甲基化(H3K79me)[1,2,3]。研究光调节的分子机制有望为通过光限制或抑制有效控制赤潮提供基础。因此,为了有效预防和控制有毒赤潮的发生,需要进一步研究阐明组蛋白修饰和其他表观遗传效应对太平洋亚历山大藻生长和产毒能力的影响。 本研究中使用的太平洋亚历山大藻来自于中国海洋遗传与育种教育部重点实验室的藻类保存培养。将正常培养条件下处于对数生长期的太平洋亚历山大藻细胞接种到含有f/2培养基的海水中,以相同的初始浓度(1000 cells mL-1)暴露于正常光照(CT组,30 μmol photon m-2 s-l)、强光(HL组,60 μmol photon m-2 s-l)下。
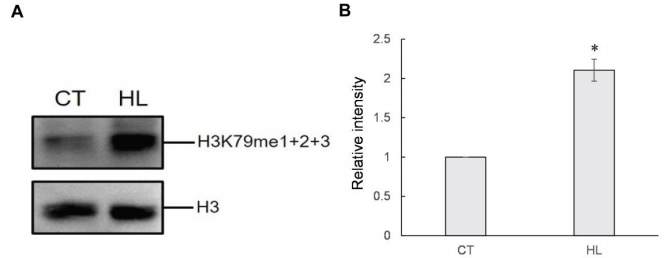
1.CT和HL条件下太平洋亚历山大藻的H3K79me丰度 首先作者利用蛋白质免疫印迹试验对HL和CT条件下的H3K79me丰度进行了分析,结果显示,与CT条件相比,在HL条件下太平洋亚历山大藻中H3K79me的丰度显著增加了2.1倍(图1)。
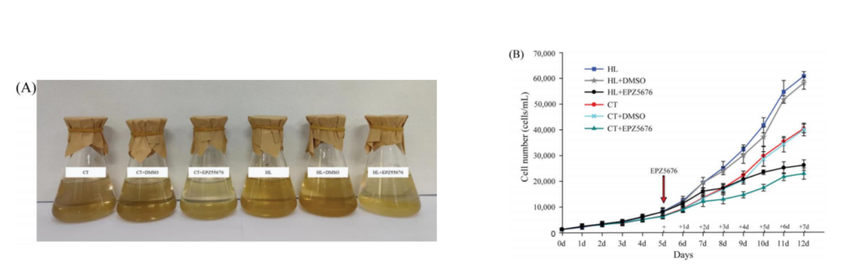
图1. 在正常光(30 μmol m-2 s -l,CT)和强光(60 μmol m-2 s -l,HL)下对太平洋亚历山大藻H3K79me进行蛋白质免疫印迹分析。 2. EPZ5676抑制太平洋亚历山大藻细胞的生长 EPZ5676是DOT1L组蛋白甲基转移酶的有效抑制剂[4],作者为了了解H3K79me修饰是否对调控亚历山大藻细胞的生长,用抑制剂EPZ5676处理太平洋亚历山大藻细胞。结果显示,在CT和HL条件下加入EPZ5676,对太平洋亚历山大藻细胞的生长均有抑制作用,且随着处理时间的延长,抑制作用更加明显。这些结果说明H3K79me参与了太平洋亚历山大藻快速生长的调控。
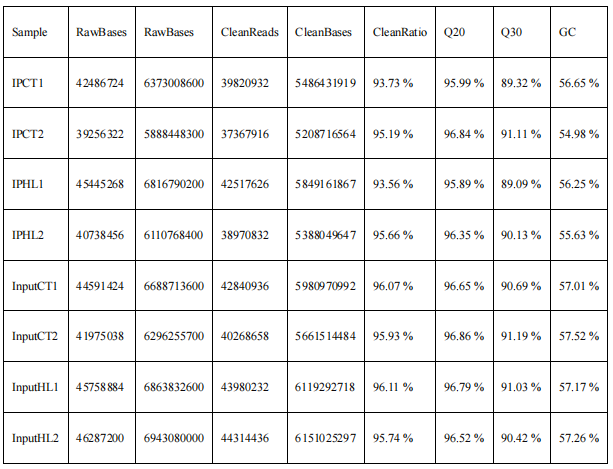
图2. 使用或不使用H3K79甲基化抑制剂EPZ5676处理的太平洋亚历山大藻的状态。 3.ChIP-seq数据与虚拟基因组的比较 为了了解H3K79me影响太平洋亚历山大藻生长的内在机制,作者利用ChIP-seq技术鉴定H3K79me相关的基因,并从可用的太平洋亚历山大藻转录组数据中生成了太平洋亚历山大藻的虚拟基因组。对HL和CT下的太平洋亚历山大藻的所有IP DNA和input DNA文库样品(每个处理包含两个生物重复)进行测序。过滤低质量数据后,每个库获得了超过3500万个Clean reads(表1)。对每个被调用的碱基进行质量评分(Q),Q20和Q30的平均值分别为95.89% - 96.86%和89.09% - 91.19%,平均GC含量(%)为56.5%,大约11.84%–15.30%的reads被比对到太平洋亚历山大藻的虚拟基因组。低map率的可能原因包括H3K79me在太平洋亚历山大藻基因的外显子区域中相对较低的分布。 表1. 8个ChIP-seq文库的测序结果汇总
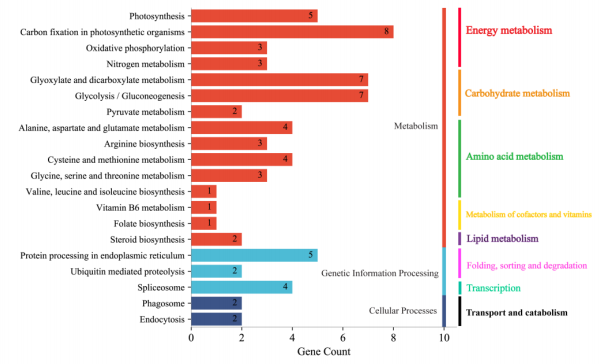
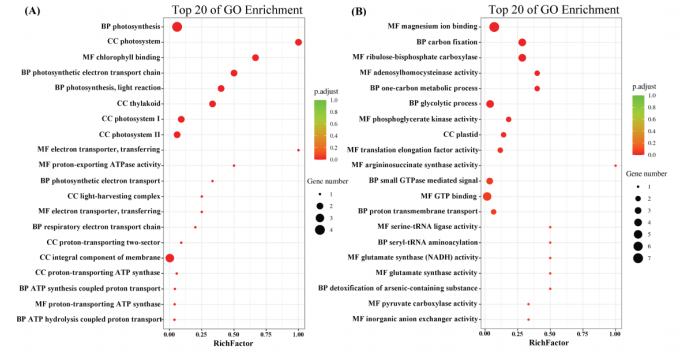
4.CT和HL条件下H3K79me的峰值分析 为了获得在HL vs. CT条件下与H3K79me相关的差异修饰基因,作者对CT和HL条件下获得的H3K79me的峰进行了分析。结果显示,CT和HL条件下分别有205和353个与H3K79me相关的峰具有特异性,而有247个峰在CT和HL条件下是共有的。在CT和HL条件下共有的247个峰中,有10个在HL条件下显著上调。 接着作者对HL条件特异性的10个峰和205个峰进一步分析发现它们与203个基因相关,对203个基因进行KEGG富集分析得到的前20个代谢通路,主要涉及 "新陈代谢"、"遗传信息处理 "和 "细胞过程"。“代谢”的类别有“碳水化合物代谢”、“氨基酸代谢”、“辅因子”、“维生素代谢”和“脂质代谢”(图3)。此外,作者还对HL和CT中太平洋亚历山大藻的差异上调峰相关基因进行了GO富集。与KEGG富集结果一致,属于代谢的“光合作用”、“碳固定”和“氨基酸合成”相关的基因被显著富集(图4)。综上所述,对ChIP-seq结果的差异富集分析表明,H3K79me主要通过光合作用、碳固定和氨基酸合成参与HL下的快速生长。
图3. HL上调峰与HL特有峰相关基因的前20个富集KEGG通路分类图
图4. HL和CT条件下太平洋亚历山大藻不同峰值相关基因的GO富集分析。 5. 通过ChIP-qPCR分析进行验证 为了证实ChIP-seq的结果,作者选取了5个上调峰关联基因CP43、PsaB、ATPase、Cyt b6/f和PSI-B进行ChIP-qPCR,以观察在不同条件下(HL和CT,以CT为参考)这些基因的编码区是否存在H3K79甲基化的富集。结果如5B所示,与CT条件相比,HL条件下与CP43、PsaB、Cyt b6/f和PSI-B基因相关的H3K79me的丰度明显增加,H3K79me的富集程度约为CT条件下的2倍。这些结果验证了ChIP-seq数据的可靠性(图5A)。
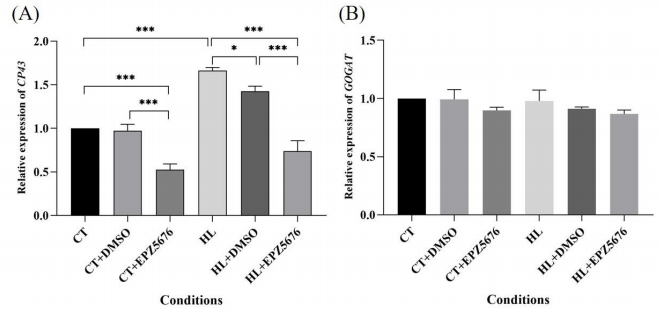
图5. 基因编码区H3K79me的富集分析。(A) ChIP-seq结果中H3K79me的倍数变化,(B)在HL与CT下的ChIP-qPCR。 6. HL vs. CT下H3K79me差异修饰基因和差异表达基因的关联分析 作者为了找到H3K79me在太平洋亚历山大藻HL vs. CT条件下快速生长中的下游关键靶基因,对差异表达基因(DEGs,登录号:PRJNA715858[5])与H3K79甲基化上调峰相关基因进行联合分析,发现了重叠基因CP43和GOGAT。 7. EPZ5676抑制H3K79me靶基因的表达 作者进一步利用H3K79me抑制剂EPZ5676药理实验研究了CP43和GOGAT是否参与H3K79me,从而调控太平洋亚历山大藻细胞的生长。结果如图6所示,CP43在HL条件下表达量明显增加,经H3K79me特异性抑制剂EPZ5676处理后,CP43的表达在HL和CT条件下均受到显著抑制,说明CP43能够参与H3K79me从而调控细胞的生长。然而,在CT和HL条件下,GOGAT的表达没有明显变化,对抑制剂EPZ5676的反应也是如此,作者推测GOGAT可能参与了生长调控,但不是在转录本上。
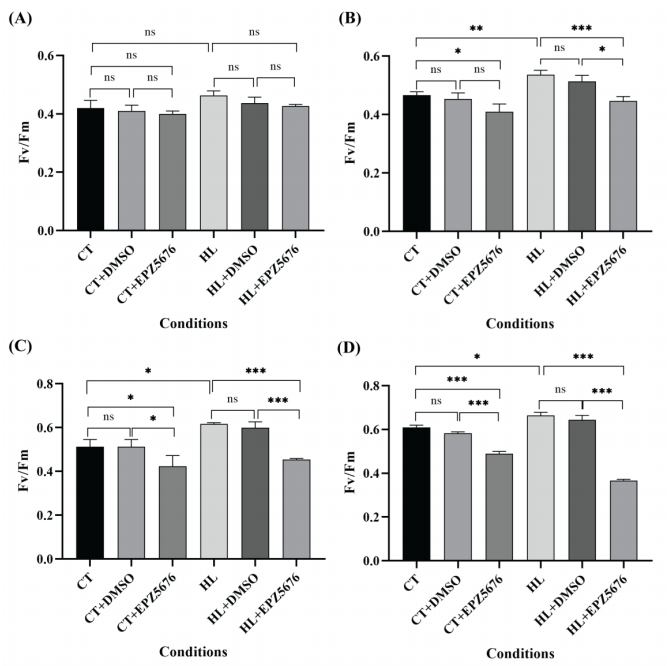
图6. 用EPZ5676 处理的太平洋亚历山大藻细胞中CP43和GOGAT的相对表达水平。 8. EPZ5676降低了太平洋亚历山大藻的最大光合能力 从上一步作者得知EPZ5676抑制了H3K79me靶基因CP43的表达,而CP43经前面ChIP-seq分析得知它参与光合作用,并且CP43可以通过蛋白水解活性快速去除光损伤过程中无法恢复的D1蛋白,促进D1蛋白的更新和光系统II的重新组装,同时抵抗光抑制作用[6],那么EPZ5676抑制剂的处理是否对太平洋亚历山大藻的光合能力产生影响。作者通过监测不同条件和抑制剂处理时间(1、3、5和7天)下太平洋亚历山大藻的叶绿素荧光参数Fv/Fm(QY_max),测定了暗适应细胞光系统II的最大量子产率(QY_max),以鉴定太平洋亚历山大藻的光合作用活性。结果如图7所示,太平洋亚历山大藻光系统II的QY_max随培养时间的延长而增加。此外,随着抑制剂EPZ5676处理持续时间的延长,对太平洋亚历山大藻最大光合能力的抑制作用也增强。因此,作者推测EPZ5676抑制剂影响了H3K79的甲基化,从而影响了太平洋亚历山大藻细胞抵抗光诱导损伤的能力,导致最大光合能力下降,对藻细胞的生长产生重要影响。
图7. 在CT和HL条件下,用抑制剂EPZ5676处理的太平洋亚历山大藻的QY_max(Fv\/Fm)随时间的变化。 作者首次利用ChIP-seq技术和基于太平洋亚历山大藻转录组构建的虚拟基因组的分析方法,探讨了H3K79me的下游效应基因。该研究结果表明,H3K79me通过光合作用调控太平洋亚历山大藻的爆发性生长,为组蛋白修饰在有害藻华形成机制中的功能提供了新的见解。
[1]Riaz, S., Niaz, Z., Khan, S., Liu, Y., Sui, Z., 2019. Detection, characterization and expression dynamics of histone proteins in the dinoflagellate Alexandrium pacificum during growth regulation. Harmful Algae 87, 101630. doi:10.1016/j.hal.2019.101630 [2]Zhu, Z., Liu, Y., Qi, J., Sui, Z., 2022. Identification of epigenetic histone modifications and analysis of histone lysine methyltransferases in Alexandrium pacificum. Harmful Algae 119, 102323. doi:10.1016/j.hal.2022.102323 [3]Zhu, Z., Qi, J., Liu, Y., Sui, Z., 2023. The H3K79 methylase DOT1, unreported in photosynthetic plants, exists in Alexandrium pacificum and participates in its growth regulation. Marine Pollution Bulletin 190, 114867. doi:10.1016/j.marpolbul.2023.114867 [4]Daigle, S. R., Olhava, E. J., Therkelsen, C. A., Basavapathruni, A., Jin, L., Boriack-Sjodin, P. A., Pollock, R. M., 2013. Potent inhibition of DOT1L as treatment of MLL-fusion leukemia. Blood 122(6), 1017-1025. doi:10.1182/blood-2013-04-497644 [5]Liu, Y., Zhu, Z., Qi, J., Sui, Z., Shang, E., Zhang, S., Niaz, Z., 2021. Comparative Transcriptome Profiling Reveals Insights Into the Mechanisms Related to Explosive Growth of Alexandrium pacificum. Frontiers in Marine Science 8:781851.doi:10.3389/fmars.2021.751851 [6]Aro, E., Virgin, I., Andersson, B., 1993. Photoinhibition of Photosystem II. Inactivation, protein damage and turnover. Biochimica et Biophysica Acta 1143, 113-134. doi:10.1016/0005-2728(93)90134-2 |